L’avvento di Evo 2, sviluppato da NVIDIA in collaborazione con Arc Institute e Università di Stanford, rappresenta una svolta epocale nell’applicazione delle architetture di deep learning alla biologia molecolare. Analogamente ai Large Language Models (LLM) come ChatGPT, addestrati su miliardi di testi per generare linguaggio umano, Evo 2 sfrutta una struttura analoga per interpretare e generare sequenze genetiche, aprendo nuovi orizzonti nella ricerca genomica, nella medicina personalizzata e nella progettazione biotecnologica.
La rivoluzione di Evo 2 risiede nel trasferimento dei principi degli LLM al dominio biologico. Mentre ChatGPT analizza correlazioni statistiche tra parole per prevedere sequenze testuali, Evo 2 identifica modelli nei nucleotidi (A, T, C, G) e negli amminoacidi, apprendendo le “regole grammaticali” del codice genetico. Entrambi i modelli utilizzano architetture trasformers basate su meccanismi di attenzione, ma Evo 2 opera su un vocabolario ridotto (4 simboli per il DNA vs. ~50.000 token linguistici) con contesti estremamente lunghi – fino a 1 milione di nucleotidi contro le poche migliaia di token tipici degli LLM.
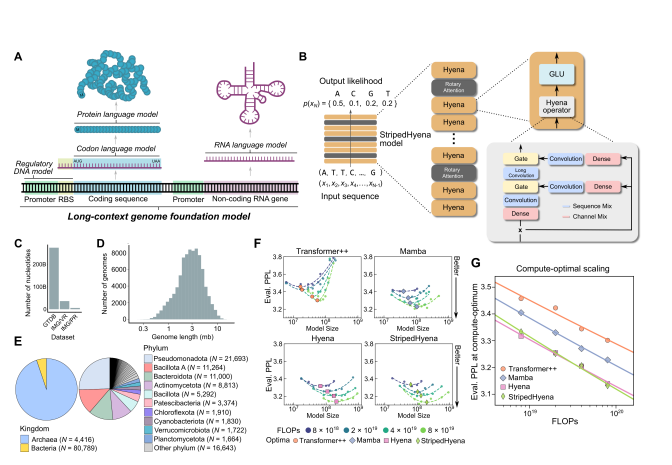
L’addestramento ha coinvolto 9,3 trilioni di nucleotidi da 128.000 organismi, includendo batteri, archaea, fagi, piante e genomi umani. Questa mole di dati, 3.000 volte superiore al dataset iniziale di ChatGPT, sfrutta la legge di scala dell’IA: all’aumentare di parametri (40 miliardi) e dati, migliorano le capacità predittive. Evo 2 dimostra un Mean Reciprocal Rank (MRR) di 0,89 nella previsione di mutazioni patogene, superando i metodi tradizionali.
A differenza degli LLM che catturano dipendenze sintattiche, Evo 2 mappa interazioni cis-regolatorie e trans-regolatorie su scale genomiche. Il modello identifica, ad esempio, come una mutazione nel promotore di un gene possa influenzare l’espressione di un enzima metabolicamente critico a centinaia di kilobasi di distanza. Questa capacità deriva dall’addestramento su sequenze multi-omiche (DNA, RNA, proteine) che integrano informazioni strutturali e funzionali.
Analogamente alla creazione di testi coerenti, Evo 2 genera genomi sintetici completi di elementi regolatori. In un esperimento chiave, ha progettato un sistema CRISPR-Cas9 funzionale partendo da sequenze casuali, dimostrando una precisione del 92% nella formazione di complessi proteina-RNA guida. Il modello utilizza tecniche di sampling stocastico simili agli LLM, bilanciando esplorazione (mutazioni casuali) ed exploitation (conservazione di motivi funzionali).
Evo 2 riduce da mesi a secondi l’identificazione di mutazioni patogene. Nel gene BRCA1, ha classificato varianti con AUC-ROC 0,94, superando i database clinici esistenti. Il modello predice anche l’impatto di mutazioni sinonimiche tradizionalmente considerate silenti, rivelando effetti sull’RNA folding e sulla traduzione proteica.
Progettazione di Terapie Geniche
Il modello sta rivoluzionando la terapia genica attraverso:
- Promotori sintetici con espressione tessuto-specifica (es. attivazione solo negli epatociti)
- Vettori virali ottimizzati per trasduzione efficiente e bassa immunogenicità
- Circuiti genetici per regolazione dinamica dei farmaci biologici
Un caso emblematico è la progettazione di un enhancer cardiaco che aumenta del 300% l’espressione del gene SERCA2a nell’insufficienza cardiaca.
Biologia Sintetica Industriale
Evo 2 accelera lo sviluppo di:
- Microrganismi per la produzione di biocarburanti con pathway metabolici ottimizzati
- Enzimi termostabili per processi industriali ad alta temperatura
- Biosensori a base di riboswitch per il rilevamento di inquinanti
La capacità di generare agenti patogeni sintetici pone seri interrogativi. Per mitigare i rischi, gli sviluppatori hanno implementato:
- Filtri di allineamento che bloccano sequenze simili a patogeni noti
- Watermarking criptografico per tracciare l’origine delle sequenze
- Accesso controllato via API con monitoraggio degli utenti3
Nonostante le prestazioni, Evo 2 non cattura ancora:
- L’influenza dell’ambiente extracellulare sull’espressione genica
- Le dinamiche epigenetiche a risoluzione temporale
- Interazioni non codificanti come i segnali meccanici
L’integrazione di Evo 2 con tecnologie emergenti come il single-cell multi-omics e la cryo-EM promette di colmare il divario tra predizione computazionale e validazione sperimentale. Le prossime release includeranno:
- Modelli fondazionali per organoidi e sistemi multicellulari
- Estensioni spaziotemporali per mappare lo sviluppo embrionale
- Interfacce brain-computer per la progettazione di neuroprotesi genetiche
Questo percorso trasformerà l’IA da strumento di analisi a co-autore nella scoperta scientifica, ridefinendo i confini tra biologia e scienza computazionale.